●「エイリアン」ナスカのミイラのゲノム配列解析は地球外起源を支持しない!

背景
9月12日、研究者達はメキシコ合衆国総会で2体のナスカのミイラを発表した。
研究者達は、これらのミイラは決定的に人間ではない(しかし人型である)生物学的実体であると報告し、ゲノム研究と解剖学的分析から得られた証拠を披露した。
もし彼らの結論が正しければ、これは生物学と分類学に対する我々の理解に重要な変化をもたらすものである。
ホセ・デ・ラ・クルス・リオス、ハイメ・モウサン、ホセ・デ・ジェス・ザルチェ・ベニテスはメキシコシティで研究結果を発表した。
3つのサンプルから得られたゲノム・リードは、公聴会で発表されたいくつかの遺伝子解析を行ったメキシコ国立自治大学所属の研究者によって、NIHシーケンス・リード・アーカイブ(SRA)に提出された。
研究者によれば、サンプルを遺伝子解析のために複数のグループに送ったが、いずれも議会での公聴会には直接関与していないようである。
Aliens 👽 mummy #aliens #alienbody pic.twitter.com/XteGYpvMqY
— World War 3 (@Worldwar_3_) September 14, 2023
ミイラに関する既知の情報:
発表者3人全員が、2018年にペルー共和国議会で、ヒト型ではないナスカのミイラに関する証拠を発表したことがある。メキシコ議会で提出された証言によれば、これらは以前に示されたものと同じミイラのサブセットであるようだ。
このミイラは、その結果を記録し、さらに詳しい情報を掲載したDVDを販売しているチームによって広範囲に調査され、www.the-alien-project.com 3本指の手と足、細長い頭蓋骨、頭蓋骨の中央にある脊椎接続部を持っている。
これらの標本についての徹底的な論証の概要は、https://antropogenez.ru/review/1119/
非網羅的に要約すると、ミイラ発見グループから独立したX線専門家は、この標本は人間の骨を生物学的に意味のない配置に意図的に配置したものであると述べている。
メキシコ会議で発表された "人型爬虫類 "タイプの細長い頭蓋骨は、真の古代ナスカのミイラと同時期にミイラ化した他の哺乳類の部分的な頭蓋骨であるという仮説がある。
メキシコ合衆国総会での発表の際、研究者たちはこれらの標本が2015年に発見されたものであり、ペルーですでに発表されている標本と同時期に発見されたものであることを示した。
メキシコ議会の標本は、エイリアン・プロジェクトのウェブサイトで発表された「人型爬虫類」タイプと識別可能である。

こちらは以前人型爬虫類と説明された標本「アルベルト」

こちらは9月12日にメキシコ議会で発表された非ヒト型ヒューマノイドの一つ。「アルベルト」に似ていることは明らかである。
古代のサンプルからの低収量抽出と一致する非アライメントリードの優勢:
提供されたSRAサンプルは、エイリアン・プロジェクトがウェブサイトにアップロードした2018年のAbraxas(アブラクサス・バイオシステムズ)のコンサルティング・レポートで議論されたサンプルと同じ塩基数、GC含量、サンプル識別子を持っている。
(編者注: SRA:Sequence Read Archive公共データーベース
これらのデータは、AbraxasのサンプルとSRAサンプルが同じであることを示している-特に同一の塩基数。
Abraxasの報告書では、サンプルAncient002(「サンプル2」)とサンプルAncient004(「サンプル4」)は、"ビクトリア "と呼ばれる同じミイラの異なる場所(骨と組織)から採取されたものであると説明されている。
「ビクトリア」は首のない人型のミイラで、メキシコ合衆国総会に提出されたものではない。
サンプルAncient003(「サンプル3」)は別の手として記述されている。
SRAの各サンプルにはBioSampleのアクセッションがあり、3つのサンプルはすべて提出者によってヒトと同定された。
サンプルAncient002(「サンプル2」)とAncient003(「サンプル3」)は骨と同定され、サンプルAncient004(「サンプル4」)は筋肉組織と同定された。
サンプルのGC含量は39.7-46.4%であり、ヒトDNAのGC含量の範囲と矛盾しない。
3検体それぞれについて、ネイティブSRA分類分析が可能である。
サンプル2のGC含量39.7%はヒトDNAとしては比較的低いが、例えばマメ科植物ではより典型的である。
サンプル2の42.89%のリードは、マメ科の植物であるPhaseolus vulgaris(インゲンマメ)であると確信できる。これは、サンプルの汚染や、豆由来の推定骨片の構造によって最も簡単に説明できる。

SRA分類学解析では、サンプル3の97.38%のリードが既知の分類学的カテゴリーに確実に割り当てられています。
ホモ・サピエンスに自信を持って割り当てられるリードは30.22%に過ぎず、これは一見、非ヒト由来のDNAの存在を示しているように思われる。しかし、これを既知の高品質ヒトサンプルのSRA分類学分析と比較してみましょう。
ここでは、93.15%のリードしか確信を持って同定できないことがわかります。
また、ホモ・サピエンスに自信を持って割り当てられたリードはわずか12.04%で、サンプル3で割り当てられた30.22%よりはるかに低い。
このことから、サンプル3はほぼ間違いなくヒトのDNAである。
前述したAbraxasの報告書でも、サンプル3はヒトのDNAを含み、さらにヒトの男性であると特定されている。

サンプル4の63.72%のリードは未同定である。これは、何らかの品質管理の問題、つまりサンプルの汚染や非常に低品質なデータに起因する可能性があると解釈するのが最も簡単である。
Abraxasのレポートでは、サンプル4のリードと既知のゲノムを照合するために行われたbioinformatics(バイオインフォマティクス)作業について述べられている。
注目すべきは、304,785,398本の重複リード(SRAにアップロードされたリードは受けていない処理ステップ)が、テストしたゲノムのいずれとも一致しなかったことである。
しかし、重複リードを除去すると、この数は10分の1の30,823,217に減少した。
(編者注:バイオインフォトマティクス:生命科学と情報科学の融合分野。DNAやRNA、蛋白質を始めとする生命がもつ様々な情報を対象に、方法論やソフトを開発し分析して解明する事)
この解析を続けて、サンプル4のユニークな未知のリードをコンティグに組み立てた。
マッピングされていないリードの65.69%がアセンブル(集める事)に成功し、NCBI ntデータベースの既知の生物に再マッチした。(編者注:NCBI:米国立バイオテクノロジー情報センター)
アセンブルされたコンティグの97%は、ntデータベースの配列とのマッチングに成功した。
要約すると、サンプル4で被検生物種にマッチできなかったリードは、平均して重複の多いリードであった。
重複を除去し、残りの未知のリードをコンティグにアセンブルすると、これらの残りの未知のリードの64%を既知の生物配列のデータベースにマッチさせることができた。
Abraxasの報告書は、NCBI ntデータベースにはすべての既知生物の配列が含まれているわけではないこと、したがって、未同定のDNAリードが、データベースに含まれていない既知の(したがって陸生の)生物のものである可能性があることを認めている。
SRAの分類学的解析の数字は、64%が未同定であることを示唆している。
しかし、これは曖昧さのない古代ヒトのDNAでも珍しいことではないことがわかる。

SRR17043540は古代マルタゲノムの研究によるもので、SRA分類学解析ではこのサンプルについて57%の未同定リードが得られている。

ERR4863252は、現在のフランスに相当する場所で採取された古代人1個体からのサンプルです。
このサンプルの大半のリードは同定されていますが、SRA分類学解析では31.27%のリードが未同定です。また、11.04%だけが確信を持ってヒトと推定された。
結論:
さて、ナスカの "エイリアン・ミイラ "を取り巻く状況と、非ヒトの証拠として提示された遺伝子データを再検討した結果、どのような結論が導き出せるだろうか?
遺伝子データが非ヒト起源を示す決定的な証拠ではないことは明らかである。
エイリアンの形態の証拠として支持されているX線証拠の問題点と合わせると、ナスカのミイラは説得力を持たない。
それらは古代の材料から作られたものかもしれないが、古代のエイリアンの遺体ではない。
●Genome sequencing analyses of “alien” Nazca mummies do not support an extraterrestrial origin
Context
On September 12, researchers presented 2 Nazca mummies to the General Congress of the United Mexican States. They report that these mummies are definitively non-human (but humanoid) biological entities and showcased evidence from genomic studies and anatomical analyses. If their conclusions are correct, this indicates an important change in our understanding of biology and taxonomy – to say nothing of the more sensationalist claims that the mummies are extraterrestrial in origin.
José de la Cruz Ríos, Jamie Maussan, and José de Jesus Zalce Benitez presented their findings in Mexico City. Genomic reads from 3 samples have been submitted to the NIH Sequence Read Archive (SRA) by a researcher affiliated with the Universidad Nacional Autónoma de México who performed some genetic analysis presented in the hearing. The researchers say they sent the samples to multiple groups for genetic analysis, none of whom appear to have been directly involved in the hearing before the Congress.
Previously known information on the mummies
All 3 of the presenters have previously presented evidence on non-human humanoid Nazca mummies before the Congress of the Republic of Peru in 2018. Based on the testimony provided before the Mexican Congress, these seem to be a subset of the same mummies previously shown. The mummies have been extensively examined by a team who documents their results and sells DVDs with further information on www.the-alien-project.com They have 3-fingered hands and feet, elongated skulls, and spinal connections in the center of the skull.
An overview of a thorough debunking of these specimens can be accessed at https://antropogenez.ru/review/1119/. In non-exhaustive summary: X-ray experts independent of the mummy discovery group say that the specimens consist of human bones intentionally placed into an arrangement which makes no biological sense. The elongated skulls on the ‘humanoid reptile’ type, presented to the Mexican Congress, are hypothesized to be partial skulls from other mummified mammals contemporaneous to true ancient Nazca mummies.
During the presentation before General Congress of the United Mexican States, the researchers indicated that these specimens were discovered in 2015, at the same time as specimens which have already been presented in Peru. The Mexican Congress specimens are identifiable as the “humanoid reptile” type presented on the alien project website.
Here is “Alberto”, a specimen previously described as a humanoid reptile.
Here is one of the non-human humanoids presented before the Mexican Congress on September 12. The resemblance to “Alberto” is clear.
Preponderance of unaligned reads consistent with low yield extractions from ancient samples
The SRA samples provided have the same base count, GC content, and sample identifiers as samples discussed in an Abraxas Biosystems consulting report from 2018, uploaded by the Alien Project on their website.These data indicate that the Abraxas samples and SRA samples are the same – particularly the identical base count. The Abraxas Biosystems report describes sample Ancient002 (“sample 2”) and sample Ancient004 (“sample 4”) as being from different locations (bone and tissue) on the same mummy, called “Victoria”. “Victoria” is a headless humanoid mummy, and not one of the ones presented to the General Congress of the United Mexican States. Sample Ancient003 (“sample 3”) is described as a separate hand.
Each sample in the SRA has a BioSample accession, and all 3 samples were identified by the submitter as human. Samples Ancient002 (“sample 2”) and Ancient003 (“sample 3”) are identified as bone, and sample Ancient004 (“sample 4”) is identified as muscle tissue. GC content of the samples ranges between 39.7-46.4%, which is not inconsistent with the range of GC content in human DNA. Native SRA taxonomy analysis is available for each of the 3 samples. Sample 2’s 39.7% GC content is relatively low for human DNA, but is more typical of legumes, for example – more on this below.
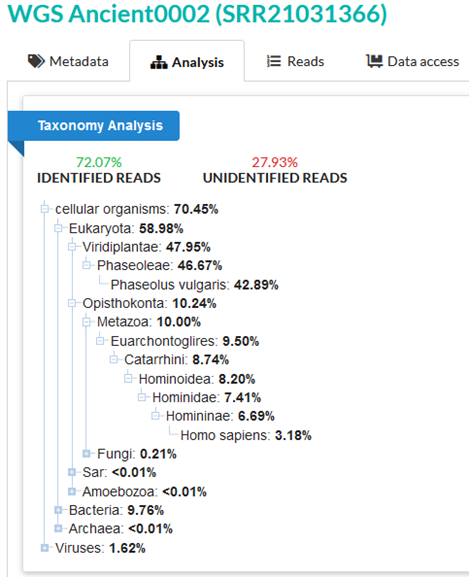
42.89% of reads in sample 2 are confidently assigned to Phaseolus vulgaris, the common bean. This is most easily explained by sample contamination or construction of the putative bone fragment from a bean derivative.
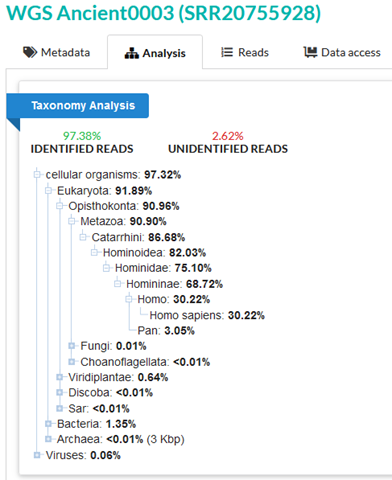
SRA taxonomy analysis confidently assigns 97.38% of the reads in sample 3 to known taxonomic categories. Only 30.22% of reads can be confidently assigned to Homo sapiens, which can initially seem like an indication of some DNA of non-human origin. However, let’s compare this to an SRA taxonomy analysis of a known high-quality human sample.

Here, we see that only 93.15% of reads can be confidently identified – this is actually lower than the percentage of identified reads in sample 3. And only 12.04% of reads are confidently assigned to Homo sapiens – much lower than the 30.22% which can be assigned in sample 3. In this context, sample 3 is almost definitively human DNA. The Abraxas report, discussed earlier, also identifies sample 3 as containing human DNA, and further specifically as a human male.
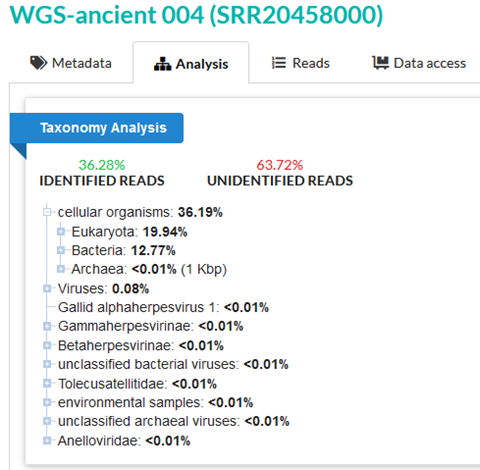
63.72% of reads in sample 4 are unidentified. This is most easily interpreted as a quality control issue of some kind – potentially caused by sample contamination, or very low-quality data.
The Abraxas report discusses the bioinformatics work that was done to match sample 4 reads to known genomes. Of note, 304,785,398 overlapped reads – a further processing step which the reads uploaded to SRA have not undergone – did not match to any of the tested genomes. However, after removing duplicate reads, this number was reduced by a factor of 10 to 30,823,217.
Continuing this analysis, they assembled the unique unknown reads for sample 4 into contigs. 65.69% of the unmapped reads were successfully assembled and re-matched to known organisms in the NCBI nt database. 97% of the assembled contigs were successfully matched to sequences in the nt database.
To summarize, the reads in sample 4 which could not be matched to tested species are on average highly duplicated reads. When duplicates were removed and the remaining unknown reads assembled into contigs, it resulted in the ability to match 64% of these remaining unknown reads to a database of known organism sequences.
The Abraxas report concludes with an acknowledgment that the NCBI nt database does not contain all sequences for all known organisms, and it is therefore certainly possible that the unidentified DNA reads are from already known (and therefore terrestrial) organisms which are not in the database.
The SRA taxonomy analysis figures still seem evocative, though – 64% unidentified? However, we can see that this is not unusual even for unambiguous ancient human DNA.
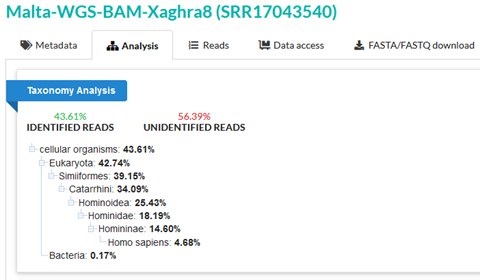
SRR17043540 is from a study into ancient Maltese genomes, and we can see that SRA taxonomy analysis gives 57% unidentified reads for this sample.
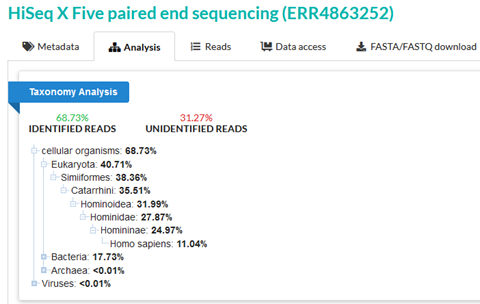
ERR4863252 is a sample from a single ancient human individual from the location corresponding to present-day France. Although the majority of reads in this sample are identified, 31.27% of reads are still unidentified by the SRA taxonomy analysis. And only 11.04% are confidently assigned as human.
Conclusion
So, after a review of the context surrounding the Nazca “alien mummies” and the genetic data presented as evidence of non-humanity – what conclusions can we draw? It seems clear that the genetic data is not conclusive evidence of non-human origins. Combined with the problems with the X-ray evidence espoused as proof of alien morphology – the Nazca mummies are not convincing. They may be assembled from ancient materials, but they are not ancient alien bodies.
Image Sources
[1] https://www.the-alien-project.com/en/mummies-of-nasca-alberto
[2] https://www.youtube.com/watch?v=7GVWup8KeQw
[3] https://trace.ncbi.nlm.nih.gov/Traces/?view=run_browser&acc=SRR21031366&display=analysis
[4] https://trace.ncbi.nlm.nih.gov/Traces/?view=run_browser&acc=SRR20755928&display=analysis
[5] https://trace.ncbi.nlm.nih.gov/Traces/?view=run_browser&acc=SRR24975192&display=analysis
[6] https://trace.ncbi.nlm.nih.gov/Traces/?view=run_browser&acc=SRR20458000&display=analysis
[7] https://trace.ncbi.nlm.nih.gov/Traces/?view=run_browser&acc=SRR17043540&display=analysis
[8] https://trace.ncbi.nlm.nih.gov/Traces/?view=run_browser&acc=ERR4863252&display=analysis
@KzUFO現象調査会

