学とみ子:以前に、TSさんが、TCRについて質問をしてきたことがありました。
以下のサイトを見つけました。
ものづくりのための研究ノート043:STAP細胞=ES細胞でない??「kahoの日記」の意外な結末!!
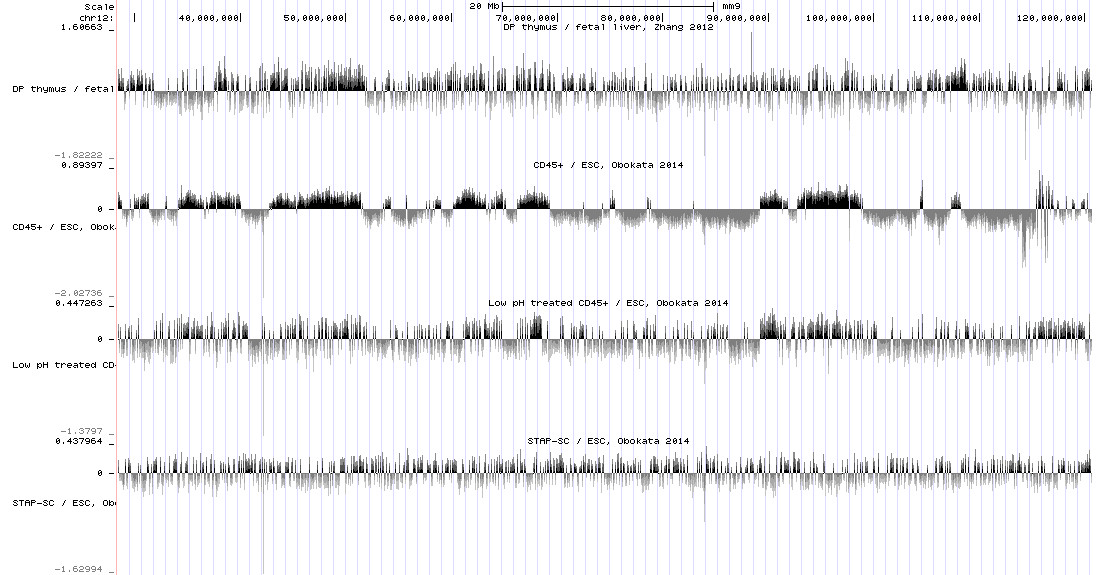
(以下の図参照 一番上:コントロール細胞(別論文の細胞)とES細胞の比較、2番目CD45+細胞とES細胞の比較、3番目STAP幹細胞とES細胞の比較。図のような染色体全体のリード数のパターンをみていると、CD45+細胞とES細胞は極端に違うこと(2番目)、またES細胞とSTAP幹細胞(4番目)は同様のパターンであることはわかるのですが、STAP細胞(3番目)に関してはCD45+細胞とES細胞もしくはSTAP幹細胞細胞の真ん中(というか足して2で割った)のような特徴があります。図でも目で見てわかる程度にパターンが違います。)。
ネットブックで行うNGS解析007:ちょいしょぼ解析覚書。。
共同研究でRNA-seqデーター解析をしており、性懲りもなくデーターをもらってきて勉強がてら解析してみることにした(*)。
https://www.ncbi.nlm.nih.gov/gene/21577
ハツカネズミのTCRβ遺伝子の場所についての情報です。参考になりますでしょうか?
別件です。
WikiPathologica の情報です。ただし、これはヒトの話です。
http://www.ft-patho.net/index.php?T-cell%20receptor(TCR)%20gene
•TCRβ遺伝子は7q34に685kbにわたり局在している。TCRγ, TCRδ遺伝子と異なり, β遺伝子のV領域はより複雑となっている。約65のVβ遺伝子エレメントが存在し、名称のつけ方には2通り(Ardenら, Weiら)がある。Vβ遺伝子は34のファミリーに分類される。異なる名称のつけ方がRowenらにより提唱され, この30のVβ遺伝子サブグループはIMGTにより採用されている。(IMGT: the international ImmunoGeneTics database-->IMGT web page)
STAP細胞(3番目)に関してはCD45+細胞とES細胞もしくはSTAP幹細胞細胞の真ん中(というか足して2で割った)のような特徴があります。
これは、CD45+細胞のTCRは、一方のアレルが再構成済であり、片方はGLパターンをとりますね。STAPは種類の異なる細胞が混じっているので、様相が異なります。ES細胞は、両アレルとも、TCR再構成とはまったくないので、STAP細胞とは異なることが示されているように思いますが、どうでしょうか?