東京大学先端科学技術研究センターの西増弘志教授らのグループとアーク研究所のパトリック・スー博士らのグループによる共同研究チームが、大腸菌の中にDNAを組み換えできる能力を持つ分子システムを発見したと発表した。この研究成果は6月26日、科学誌「Nature」に2報同時掲載された。
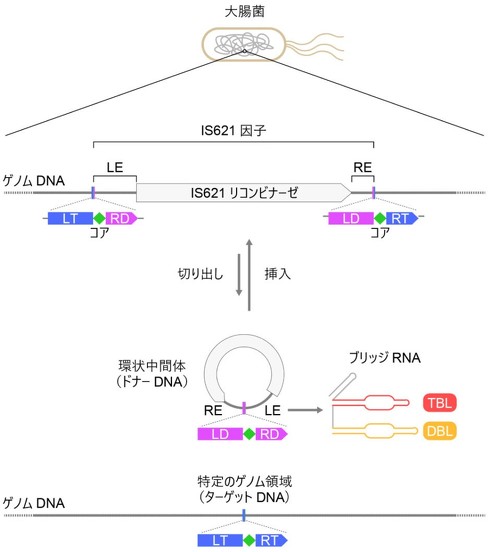
研究チームは、大腸菌のゲノムに存在する「IS621転移因子」が2つの重要な要素を産生することを明らかにした。1つは「IS621リコンビナーゼ」と呼ばれるタンパク質性の酵素であり、もう1つは「ブリッジRNA」と名付けられた非コードRNAである。これらの分子が協力して働くことで、DNAの特定の部分を切り取り、別の場所に組み換える複雑な過程を実現する。このシステムの仕組みは以下の通りである。まず、IS621リコンビナーゼがブリッジRNAと複合体を形成する。次に、この複合体がドナーDNA(移動させたいDNA)とターゲットDNA(移動先のDNA)の両方に結合する。その後、IS621リコンビナーゼがDNAの切断や交換、再結合という複雑な過程を段階的に行う。
IS621リコンビナーゼの特筆すべき点は、その「プログラム可能性」にある。通常、DNA組換え酵素は特定のDNA配列しか認識できないが、IS621リコンビナーゼはブリッジRNAの助けを借りることで、さまざまなDNA配列を認識し、編集ができる。ブリッジRNAはドナー結合ループ(DBL)とターゲット結合ループ(TBL)という2つの重要な領域を持っており、DBLがドナーDNAと、TBLがターゲットDNAと相補的に結合する。これらのループのDNA結合領域の塩基配列を変更できることで、さまざまな塩基配列をもつドナーDNAとターゲットDNAの間の組換えが可能に。IS621リコンビナーゼは「プログラム可能な」RNA依存性DNA組換え酵素として機能する。
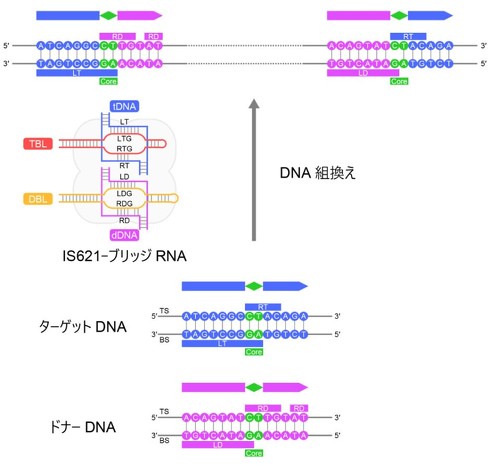 IS621リコンビナーゼとブリッジRNAが触媒するDNA組換え反応
IS621リコンビナーゼとブリッジRNAが触媒するDNA組換え反応
研究チームは、クライオ電子顕微鏡を用いてこの分子システムの詳細な立体構造を解明した。その結果、4分子のIS621リコンビナーゼ、ブリッジRNA、ドナーDNA、ターゲットDNAが複雑に絡み合った構造が明らかになった。この構造解析により、DNA編集の詳細なメカニズムを解明できた。
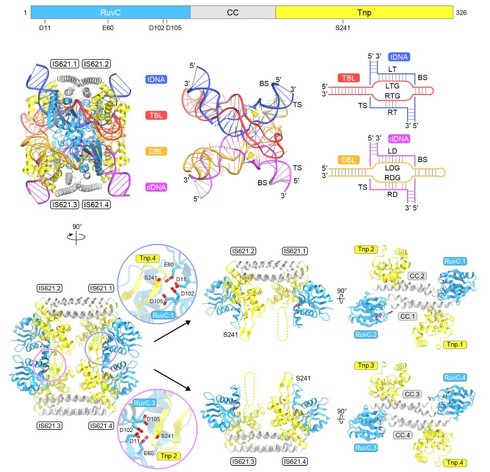 IS621リコンビナーゼ、ブリッジRNA、ドナーDNA、ターゲットDNA複合体の立体構造
IS621リコンビナーゼ、ブリッジRNA、ドナーDNA、ターゲットDNA複合体の立体構造
この新しいDNA編集システムは、既存のCRISPR-Cas9システムとは異なる特徴を持っている。Cas9は1か所のガイド領域を持つガイドRNAを使用し、1つのターゲットDNAを認識して切断。一方、IS621は4か所のガイド領域を持つブリッジRNAを使用し、ターゲットDNAとドナーDNAの2つを同時に認識する。また、IS621はDNAの切断だけでなく、交換・再結合も行う複雑な反応を触媒する。この特性により、IS621システムはCRISPR-Cas9では困難だった長鎖DNAのノックインなど、より複雑なゲノム改変ができる可能性がある。そのため、IS621システムは次世代のゲノム編集ツールとして期待されている。